| 设为首页 | 加入收藏 |
|
English |

近日,中国科学院中-非联合研究中心依托单位中国科学院武汉植物园万涛和王青锋研究员团队在Plant Communications在线发表了题为“The origin, evolution and diversification of MADS-box transcription factors in green plants”的研究论文。该研究重建了绿色植物MADS-box转录因子家族的演化历史,提出植物Type I基因多系起源的假说,确定陆生植物MIKCC和MIKC*基因的分化发生链型植物共同祖先,并提出了种子植物MIKCC基因的渐进式演化模型,为深入认识该类转录因子的演化和功能分化提供了重要见解。
研究背景
MADS-box基因是一类在真核生物中广泛分布的转录因子基因家族,其编码的蛋白含有保守的MADS结构域(M结构域),可以同源或异源二聚体的形式结合靶基因启动子上的CArG-box(C-A-rich-G-box)元件,参与真核生物多种关键发育过程的调控。传统观点认为,MADS-box家族Type I(植物中称Type I,其他真核生物中称SRF-type)和Type II(植物中称Type II,其他真核生物中称MEF2-type)两类基因的分化源于真核生物共同祖先的一次基因复制。但近期研究提出,植物Type I和Type II基因源于MEF2-type祖先基因在陆生植物共同祖先中的基因复制,对植物Type I基因是SRF-type祖先基因起源的经典假说提出了挑战。陆生植物Type II基因也称MIKC型基因,编码M(MADS)、I(Intervening)、K(Keratin-like)和C(C-terminal)结构域。早期研究认为,MIKC基因起源于链型植物(轮藻植物和陆生植物),并在陆生植物中进一步分化为MIKCC和MIKC*两类。然而,陆生植物MIKCC和MIKC*基因的具体分化时间仍存在争议。MIKCC类蛋白广泛参与被子植物的花器官四聚体复合物(FQC,Floral Quartet Complex)组装,其多样化被认为是开花植物花器官形态演化的重要驱动力之一。MIKCC蛋白如何演化获得形成四聚体的能力,仍是该类基因演化和功能分化研究中的关键科学问题之一。
研究内容
本研究整合了覆盖真核生物主要谱系的551个代表性物种的基因组和转录组数据,系统鉴定了MADS-box家族的成员,通过使用多种进化树构建策略,结合正选择分析、同源搜索、多序列比对、蛋白结构预测、保守结构与分析以及内含子-外显子分析,系统探讨了MADS-box家族基因在绿色植物中的起源、扩张与功能演化。
(1)真核生物MADS-box基因的多样化分布
从445个物种中鉴定到4,899条序列,陆生植物MADS-box家族成员数量显著高于其他真核谱系,并从苔藓植物到被子植物呈递增分布趋势,表明该家族在陆生植物中经历了大规模扩张,且扩张过程与植物陆地化和陆生植物辐射演化密切相关(图1A)。植物的绝大多数MADS-box基因经历了纯化选择(dN/dS (ω) < 1),且链型植物Type II基因经历了更强的选择压力(图1B)。陆生植物Type I基因比Type II基因具有更高的ω值,一方面反映Type I基因可能经历了更高的birth-and-death速率,另一方面表明Type II基因在陆生植物演化过程中可能扮演着重要角色。
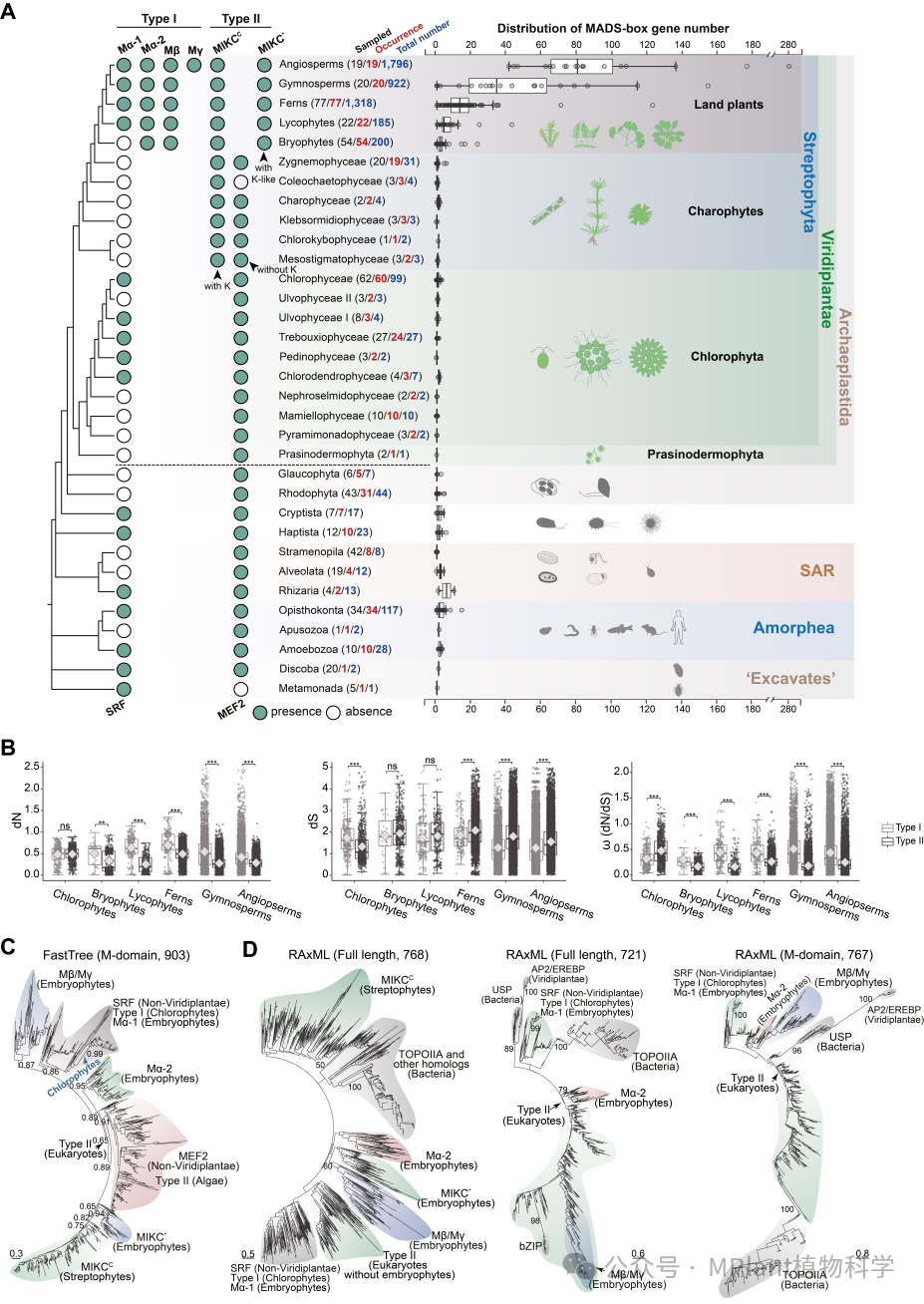
图1. 真核生物MADS-box基因的分布及其演化
(2)绿色植物Type I基因的多系起源
陆生植物Type I基因由Mα、Mβ和Mγ三类组成。研究结果表明,陆生植物Mα基因可被分为Mα-1和Mα-2两类,其中Mα-1基因与所有SRF-type基因和绿藻Type I基因直系同源,Mα-2基因与陆生植物Mβ/γ基因、真核生物Type II基因直系同源(图1C-D、图2)。因此,本研究提出绿色植物MADS-box家族Type I基因是多系起源的假说,即绿藻Type I和陆生植物Mα-1基因起源于SRF-type祖先基因,陆生植物Mα-2、Mβ和Mγ基因起源于MEF2-type祖先基因。
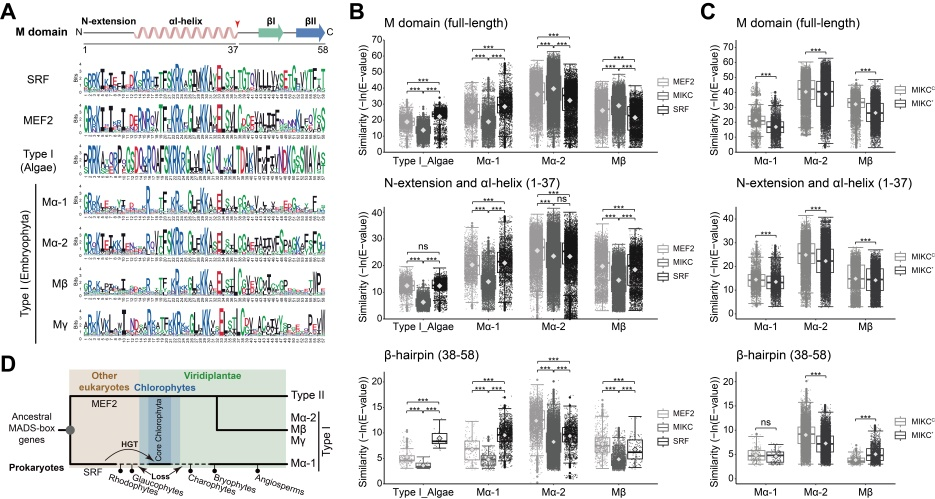
图2. 绿色植物Type I基因的多系起源
(3)链型植物古老基因复制事件塑造了Type II基因的早期演化
进化分析结果表明,Type II基因在链型植物共同祖先物种中可能经历了两次复制,第一次复制产生了编码和不编码K结构域的两类基因,第二次复制导致了K结构域编码基因分化为MIKCC和MIKC*两类,并且MIKC*的直系同源基因在轮藻中丢失(图3A)。进一步分析证实,相比于陆生植物MIKC*基因,轮藻MIKC和陆生植物MIKCC基因具有更近的亲缘关系(图3A-E)。同义替换速率(Ks)推断表明,MIKCC与MIKC*的分化时间早于轮藻植物和陆生植物谱系的分歧时间(图3F)。综上,本研究提出将轮藻植物MIKC基因重新归类为MIKCC基因,陆生植物MIKCC和MIKC*基因的分化是由链型植物共同祖先中的基因复制引起,而非陆生植物共同祖先中的基因复制(图3G)。
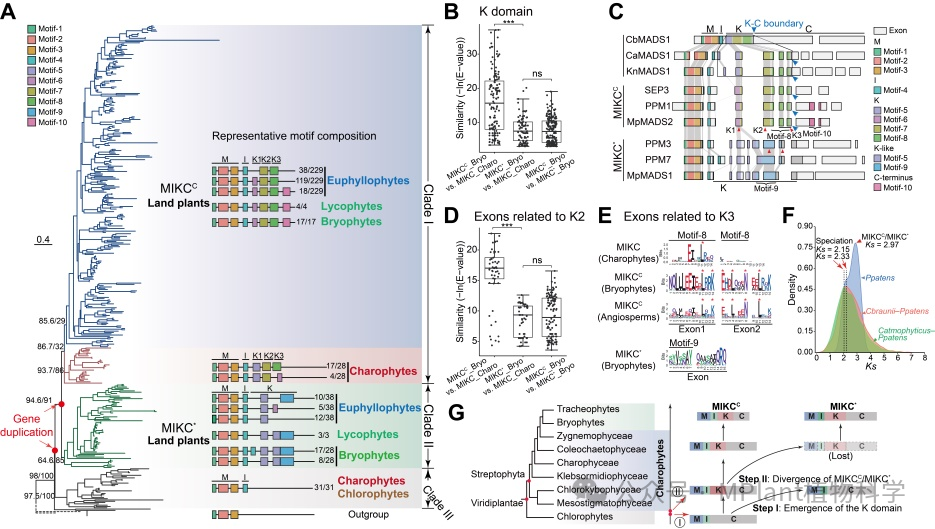
图3. 陆生植物Type II基因的早期演化
(4)渐进式演化驱动了种子植物MIKCC基因的功能多样性
研究表明,链型植物MIKCC蛋白具有球状祖先结构,其较长、无序的C-端结构将靠近N-端的M、K等关键功能结构域包裹在内,限制了蛋白四聚体复合物的形成;而基因组重排导致外显子断裂,使陆生植物MIKCC蛋白C-端结构缩短,暴露出蛋白关键功能域,从而促进了四聚体复合物的形成,为该类蛋白后续的功能多样化提供结构基础(图3C、4A-B)。K结构域K3区疏水氨基酸残基比例在陆生植物谱系中不断上升,在蕨类植物共同祖先中达到最高,为种子植物MIKCC蛋白四聚体形成提供了化学基础(图4C)。进一步分析表明,MIKCC基因在蕨类植物共同祖先中已经分化形成四个主要基因谱系,为该类基因在种子植物中的进一步扩张和功能多样化演化奠定基础(图4D)。综上,本研究提出种子植物MIKCC基因的渐进式演化模型。
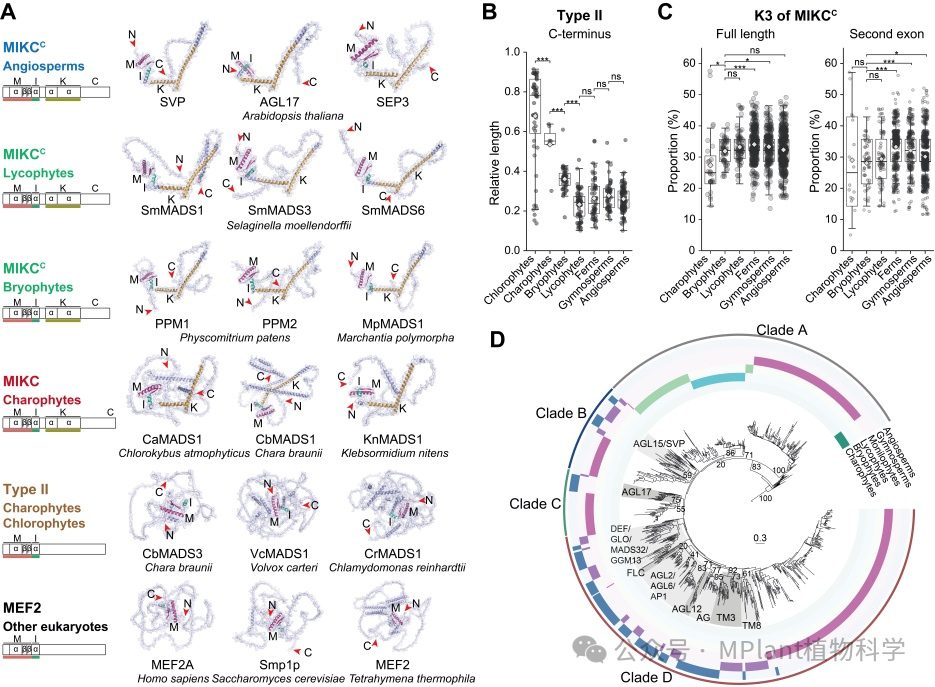
图4. 陆生植物MIKCC基因的渐进式演化
综上所述,本研究重建了绿色植物MADS-box家族基因的演化历史,提出植物Type I基因多系起源的假说,将轮藻MIKC基因重新定义为MIKCC类基因,发现链型植物共同祖先物种中的基因复制是导致陆生植物MIKCC和MIKC*基因分化的原因,MIKCC类蛋白结构和序列的渐进式演化促进其功能多样化。本研究为深入研究绿色植物MADS-box家族基因演化和功能分化奠定基础。
研究团队
中国科学院武汉植物园韩家鹏博士和万俊男博士为论文共同第一作者,万涛研究员和王青锋研究员为共同通讯作者。中国科学院武汉植物园关志林博士和许韩博士参与了该研究。该研究得到了国家自然科学基金面上项目和中国科学院中-非联合研究中心重点部署项目等课题的资助。
版权所有:中国科学院中非联合研究中心 中国·武汉 邮编:430074
电话:+86-27-87700875 电子邮件:sinafrica@wbgcas.cn
技术支持:中国科学院武汉植物园科技支撑中心 鄂ICP备05004779号-1 ![]()